i. Cree un script T5iniciales.R en R-Studio
ii. Ejecute la función data() desde la plataforma R-STUDIO e importe los conjuntos de datos trees
y npk de R; luego cree dos nuevos objetos data.frame df.arboles y df.npk; y traduzca el nombre
las columnas (variables) de ambos objetos.
iii. Para cada data frame creado, realice un análisis exploratorio de las variables respuesta (boxplots,
histogramas, frecuencias, summary, etc) considere los conjuntos de datos completos. Exporte sus
gráficas.
Para el conjunto de datos datos.npk,
iv. Calcule las medias y desviaciones estándar por bloque; cree dos subconjuntos de datos, uno para
todos los bloques pares y otro para todos los bloques nones.
Para el conjunto de datos datos.arboles,
v. Grafique por pares de variables; efectúe regresiones lineales entre el volumen (variable
dependiente) y (altura, perímetro) variables independientes; grafique los valores predichos
(ajustados) y los valores observados, exporte sus gráficas.
vi. Exporte los conjuntos de datos, datos.arboles y datos.npk a archivos .csv
vii. Exporte los valores predichos, observados y los residuales da cada modelo a un archive .csv.
viii. Genere 40 datos de peso de borregos con una media de 24 kg y sd de 5 kg (asuma normalidad).
Genere 40 datos de diámetro de cintura de borregos en cm con media 45 cm y sd 8 cm (asuma
normalidad). Grafique los datos peso (variable respuesta) vs diametro (variable predictora).
Efectúe una regresión lineal, analice y guarde sus resultados en archivos en forma ordenada y
comente sus resultados.
ix. Cree 3 pares de objetos tmin1, tmax1, tmin2, tmax2, tmax3, tmin3, de longitud 6, donde tmin son
temperaturas mínimas diarias y tmax son temperaturas máximas diarias. Luego utilice la función
tpromedio descrita en script de clase para calcular los promedios de temperatura tp1, tp2, tp3,
utilice el ciclo for para hacer el cálculo en forma iterativa.
x. Exporte a un archivo .RData el área de trabajo completa.
xi. Comente sus resultados en general.
#recupera algunas bases de datos y las hace objeto
data()
data(trees)
df.arboles <- trees
data(npk)
df.npk <- npk
#traduce los nombres de las columnas en cada df
names(df.arboles) <- c("perimetro", "altura", "volumen")
names(df.npk) <- c("bloque", "N", "P", "K", "campo")
#analisis exploratorio del df de arboles
cor(df.arboles$perimetro, df.arboles$volumen)
plot(df.arboles$volumen ~ df.arboles$perimetro, xlab = "Perimetro",
ylab = "Volumen", main = "Volumen de los arboles respecto a su perimetro")
hist(x = df.arboles$altura, xlab = "Altura", ylab = "Frecuencia",
main = "Frecuencia de arboles de acuerdo a su altura")
summary(df.arboles)
#analisis exploratorio del df de npk
boxplot(df.npk$campo ~ df.npk$bloque, xlab = "Bloque",
ylab = "Campo", main = "Comportamiento del campo con diferentes tratamientos de NPK")
hist(x = df.npk$campo, xlab = "Campo", ylab = "Frecuencia",
main = "Frecuencia del comportamiento del campo ante diferentes tratamientos")
summary(df.npk)
#para el conjunto de datos npk se calcula la media y la des. estandar por bloques
#ademas se obtienen subconjuntos de los bloques pares e impares
df.npk.media <- tapply(df.npk$campo, df.npk$bloque, mean)
df.npk.media
df.npk.sd <- tapply(df.npk$campo, df.npk$bloque, sd)
df.npk.sd
df.npk.subset.par <- subset(df.npk, df.npk$bloque == 2 | df.npk$bloque == 4 | df.npk$bloque == 6)
df.npk.subset.par
df.npk.subset.nones <- subset(df.npk, df.npk$bloque == 1 | df.npk$bloque == 3 | df.npk$bloque == 5)
df.npk.subset.nones
#para el conjunto de datos de arboles se obtienen regresiones lineales y graficas
df.arboles.reg1 <- lm(df.arboles$volumen ~ df.arboles$perimetro)
df.arboles.reg1
summary(df.arboles.reg1)
df.arboles.reg2 <- lm(df.arboles$volumen ~ df.arboles$altura)
df.arboles.reg2
summary(df.arboles.reg2)
plot(df.arboles$volumen ~ df.arboles$perimetro, xlab = "Perimetro", ylab = "Volumen",
main = "Volumen de los arboles respecto a su perimetro")
plot(df.arboles$volumen ~ df.arboles$altura, xlab = "Altura", ylab = "Volumen",
main = "Volumen de los arboles respecto a su altura")
#exporta los df creados
write.csv(df.arboles, file = "T5HJBVdf.arboles.csv")
write.csv(df.npk, file = "T5HJBVdf.npk.csv")
#crea objetos a partir de los modelos hechos y los exporta
df.arboles.mod1 <- cbind(df.arboles$volumen, df.arboles$perimetro, df.arboles.reg1$residuals)
df.arboles.mod1
df.arboles.mod2 <- cbind(df.arboles$volumen, df.arboles$altura, df.arboles.reg2$residuals)
df.arboles.mod2
write.csv(df.arboles.mod1, file = "T5HJBVdf.arboles.mod1.csv")
write.csv(df.arboles.mod2, file = "T5HJBVdf.arboles.mod2.csv")
#analisis de algunas variables de borregos
peso.bor <- rnorm(40, mean = 24, sd = 5)
peso.bor
cintura.bor <- rnorm(40, mean = 45, sd = 8)
cintura.bor
plot(peso.bor ~ cintura.bor, xlab = "Diametro", ylab = "Peso",
main = "Peso de borregos de acuerdo a su diametro")
bor.reg <- lm(peso.bor ~ cintura.bor)
bor.reg
summary(bor.reg)
#analisis de temperaturas
i <- NULL; tp1 <- NULL; suma1 <- NULL
for(i in 1:6){
tmin1 <- rnorm(1, mean = 14, sd = 2)
tmax1 <- rnorm(1, mean = 27, sd = 2)
tp1 <- (tmin1 + tmax1)/2;
suma1 <- c(suma1, tp1);
}
suma1
tp1 <- sum(suma1)/6
tp1
i <- NULL; tp2 <- NULL; suma2 <- NULL
for(i in 1:6){
tmin2 <- rnorm(1, mean = 16, sd = 2)
tmax2 <- rnorm(1, mean = 29, sd = 2)
tp2 <- (tmin2 + tmax2)/2;
suma2 <- c(suma2, tp2);
}
suma2
tp2 <- sum(suma2)/6
tp2
i <- NULL; tp3 <- NULL; suma3 <- NULL
for(i in 1:6){
tmin3 <- rnorm(1, mean = 16, sd = 2)
tmax3 <- rnorm(1, mean = 29, sd = 2)
tp3 <- (tmin3 + tmax3)/2;
suma3 <- c(suma3, tp3);
}
suma3
tp3 <- sum(suma3)/6
tp3
·
Análisis
exploratorio
En el análisis exploratorio
del conjunto de datos sobre árboles se obtuvieron las gráficas de la figura 1 y
la figura 2. En la figura 1 se ve la relación que tienen las variables
perímetro y volumen, que son directamente proporcional.
Figura 1. Relación entre las variables perímetro
y volumen. Fuente: propia
En la figura 2 se puede
apreciar un histograma de la variable altura de los árboles. Los arboles con
altura entre 75 y 80 unidades se distinguen por ser lo más numerosos.
Figura 2. Histograma de la variable altura de
los árboles. Fuente: propia.
Por el otro lado, el análisis
exploratorio de los datos sobre NPK se ilustran en las gráficas de las figuras
3 y 4. En la figura 3, se aprecia un boxplot entre las variables bloque y
campo. Se observa un mejor rendimiento en el bloque 3 por encima de los otros
bloques.
Figura 3. Boxplot de las variables bloque y
campo. Fuente: propia.
En la figura 4 se ilustra el
histograma de la variable campo. Se aprecia que el mejor rendimiento se dio
entre 55 a 60 unidades.
Figura 4. Histograma de la variable campo.
Fuente: propia.
·
Regresiones lineales entre las variables de los datos sobre
arboles
Como
se comentó con anterioridad, el perímetro y el volumen de los arboles tienen
una relación directamente proporcional con un R2 de 0.93. El modelo
que representa esta relación es:
y=5.066x-36.943
Figura 5. Regresión lineal entre las variables
volumen y perímetro. Fuente: propia.
La
relación entre la altura y el volumen de los arboles no es muy alta con un R2
de 0.34. El modelo que representa esta relación es:
y=1.543x-87.124
Figura 6. Regresión lineal entre las variables
volumen y altura. Fuente: propia.
·
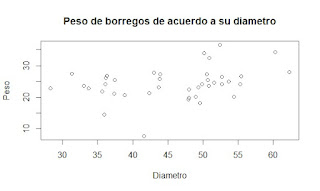
Análisis de los datos de cintura y peso de borregos
En
la figura 7 se observa la gráfica de los datos del diámetro de la cintura de
borregos con su respectivo peso. Estos datos se obtuvieron al azar de acuerdo a
algunos criterios de media y desviación estándar. Se observa que los datos no
tienen una relación con un R2 de 0.07. El modelo que representa esta
relación es: y=0.1933x+15.4369
Figura 7. Regresión lineal entre la cintura y el
peso de borregos. Fuente: propia.






No hay comentarios.:
Publicar un comentario